
Actividad no 2 con
el ADN de la Familia Blackett
Métodos de análisis de STR
Supondremos que Ud. posee el conocimiento básico de la reacción
en cadena de la polimerasa (PCR) y de la electroforesis en gel, en particular
en lo aplicable al análisis de secuencia de ADN. Nos centraremos
en las características especiales de la PCR y de la electroforesis
en gel que son relevantes para la caracterización de los STR. Si
no está Ud. familiarizado con estas técnicas, aún
debería ser capaz de completar esta Actividad.
Métodos para el análisis de los 13
loci STR del CODIS
1. Extracción del ADN
Se puede extraer ADN de casi cualquier tejido humano. Para
los ensayos de paternidad lo más habitual es usar células
bucales del interior de la mejilla. Otras fuentes de ADN procedente de
la escena de un delito pueden ser sangre, semen, tejido de una víctima
mortal, células del folículo capilar o incluso saliva. El
ADN extraído de las pruebas policiales se comparará con el
ADN extraído de muestras de referencia, de individuos conocidos.
2. Amplificación por PCR
Los cebadores de ADN se han optimizado para permitir la amplificación
de múltiples loci STR en una sola mezcla de reacción. Ajustando
cuidadosamente la distancia desde los cebadores hasta la secuencia tetramérica
repetitiva, los productos de diferentes loci no se solaparán durante
la electroforesis en gel.
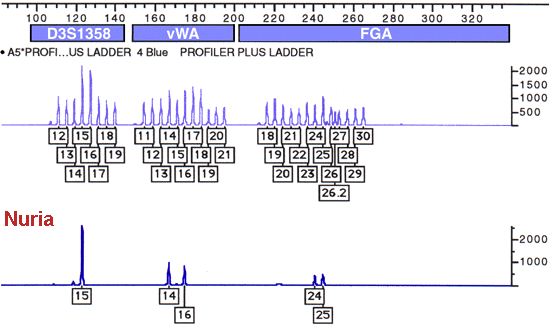 |
En los resultados parciales que se muestran arriba, se están
analizando al mismo tiempo los tres STR (D3S1358, vWA y FGA). Las longitudes
de los ADN amplificados se muestran mediante la escala de 100 a 320 pb
de la parte superior de la figura. El área central, con múltiples
picos, corresponde a patrones de referencia que contienen los alelos conocidos
para cada locus STR. Observe que los alelos de los tres loci diferentes
no se solapan. El área inferior muestra los alelos de la madre de
Bob Blackett, Nuria, para los loci D3S1358, vWA y FGA. Los alelos de Nuria
se han comparado con los patrones de referencias mediante ordenador y están
rotulados. La interpretación de este resultado indica que el
genotipo de Nuria es (15,15) en el locus D3S1358, (14,16) en el vWA y (24,25)
en el FGA.
3. Detección de los ADN tras la amplificación
por PCR
Los cebadores de PCR en los preparados comerciales (kits)
empleados para el análisis de STR tienen moléculas fluorescentes
unidas de forma covalente al cebador. Para ampliar el número de
loci diferentes que se pueden analizar en una sola reacción de PCR,
se usan múltiples conjuntos de cebadores con marcadores fluorescentes
de diferente "color". Tras la reacción de PCR, se añaden
a la mezcla de reacción patrones de longitud de ADN internos y se
separan los ADN según su longitud en un aparato de electroforesis
capilar. Los picos de ADN se van detectando según salen del gel,
mediante activación por láser. Los aparatos de secunciación
que se usan para separar los alelos y detectarlos son del mismo tipo que
los que se usan actualmente en el Proyecto de Secuenciación del
Genoma Humano, con una salida de datos digital que se puede analizar mediante
programas de ordenador específicos.
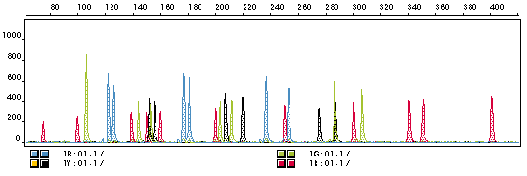 |
El producto comercial que utiliza Bob Blackett para la amplificación
por PCR se denomina AmpFLSTR™ Profiler
Plus™ PCR Amplification Kit, de la casa Applied Biosystems.
En él se analizan nueve STR empleando tres conjuntos de cebadores,
cada uno con un marcador fluorescente de distinto color. En la figura de
arriba se representan los picos de fluorescencia
azul obtenidos para 3 de los STR, fluorescencia
verde para otros 3 y fluorescencia
amarilla (mostrado en negro) para otros 3.
Los picos en rojo son los patrones
de tamaño de ADN. Mediante un programa de ordenador específico
se muestran los diferentes colores en secciones separadas de datos y se
determina la longitud exacta de los ADN. Un décimo marcador, denominado
AMEL, se usa para diferenciar el ADN de los varones (X, Y) y de las mujeres
(X, X).
En una segunda reacción de PCR se utiliza otro producto,
llamado Cofiler Plus, para amplificar otros 4 loci STR adicionales
y para repetir algunos de los loci analizados con Profiler. El resultado
de las dos reacciones de PCR constituye, por tanto, el análisis
del conjunto completo de los 13 STR del CODIS (incluyendo redundancia de
algunos loci) y un ensayo de los cromosomas sexuales. Los resultados se
obtienen en forma de alelos discretos, digitalizados, determinados a partir
del tamaño exacto de los productos amplificados por comparación
con los patrones.
|


