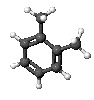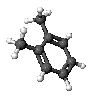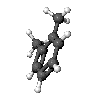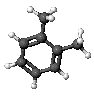
1. Dibuja aquí tu molécula en 2D:
usando el menú contextual
(ayuda)
nombre de la molécula o código PDB:
más rapidez más calidad
2. Ponle un nombre:
3. al panel derecho (se añadirán los H)
4. la estructura en 3D. (Puede ser preciso varias veces; ayuda)
5. Observa la molécula en 3D a la derecha.
6. También puedes obtener estructuras 3D escribiendo su nombre en inglés: y después puedes , por ejemplo para añadirle modificaciones.